富集表格¶
富集表格核心应用提供了通过使用 g:Profiler 的网站服务 对加载到 Cytoscape 中的任何网络的富集分析功能。单击 Enrichment Table 面板中的 Perform Gene Enrichment 按钮  或使用
或使用 Tools -> Enrichment Table -> Perform Gene Enrichment 菜单项来获取富集分析结果。
生物体和基因 ID¶
运行富集分析需要两个必要参数:
Organism:与查询基因相关的生物体Gene ID:包含基因符号的节点表列

如果有足够的可用信息,两个参数均可以由 Enrichment Table 应用预测。
- 在启动时通过处理网络中
species、organism、IntAct::species列的数据对可能的organism进行预测。 - 在启动时对
gene id列通过如下方式预测:- 从通用网络样式中检索
NODE_LABEL。 - 为
stringapp网络选择display name。
- 从通用网络样式中检索
如果你想手动设置生物体和基因 ID 或者预测不正确,可以通过单击 Enrichment Table 面板中的齿轮按钮  更改
更改 Network-specific enrichment panel settings 中的选项。
运行富集分析¶
可以通过 3 种方式运行富集分析:
- 使用
Tools -> Enrichment Table -> Perform Gene Enrichment菜单项。 - 在 Cytoscape 的
Command Line中输入enrichment analysis并回车。 - 单击
Enrichment Table面板中的Perform Gene Enrichment按钮 。
。
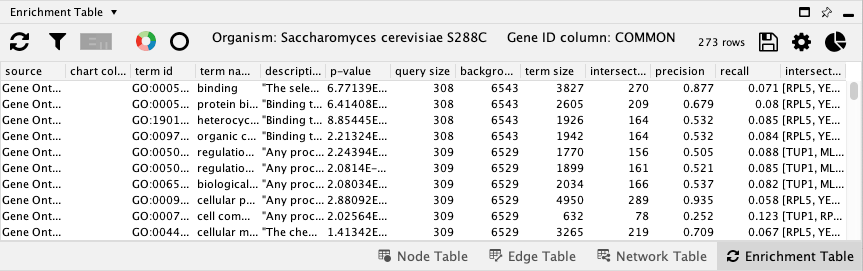
注意
默认情况下,以基因组为背景对当前网络的所有节点进行富集分析。如果选择了节点的子集,则仅使用完整网络作为背景对那些节点执行富集分析。
之后即可获得包含富集结果的表格。默认情况下,结果按照 \(p\) 值升序排列。
过滤结果¶
可以根据如下参数按类别和证据码过滤表格:
- 单击
Filter enrichment table按钮 使用过滤器。
使用过滤器。 - 在类别中选择
Gene Ontology Biological Process,单击OK。之后将得到一个过滤后的表格,表格顶部显示已过滤的行数和所有行数的摘要。 - 再次打开过滤器面板并勾选
Remove redundant terms。单击OK查看应用两个过滤器后的结果。
图表¶
图表提供了可视化网络中每个节点一组项的能力。默认情况下会显示一个圆环图,对应前 5 个项。通过最右边的 Network-specific chart settings 按钮  更改项数量和图表类型。
更改项数量和图表类型。
- 单击
Draw chart using default color palette按钮 使用默认配置创建图表。
使用默认配置创建图表。 - 单击
Reset charts删除图表。 - 单击
Network-specific chart settings更改图表设置。
项选择¶
富集表格显示基于网络中所选节点的结果。如果未选择任何节点,则所有项均显示在表格中。选择一组节点时,将显示所有选定节点的项。同样,要可视化一个或多个富集项,请选择表格中的行,相应的节点将在网络中高亮显示。
可选配置¶
除了 Organism 和 Gene ID 列外,还可以通过更改其他参数来获取更精确的富集结果。这些选项在 Network-specific enrichment panel settings  中。例如,可以通过选择适当的冗余(Jaccard)截断值(默认为 0.5)来删除表中的冗余项。
中。例如,可以通过选择适当的冗余(Jaccard)截断值(默认为 0.5)来删除表中的冗余项。
Enrichment Map 生成¶
可以根据富集表格应用生成富集数据进而生成 Enrichment Map。这需要在 Cytoscape 中安装 Enrichment Map 应用。一旦 Enrichment Map 可用,单击 Enrichment Map 图表,指定输出文件名和关联截断,单击 OK 继续。
导出结果¶
富集表格应用提供将数据导出为 csv 格式表格的能力:
- 单击
Export enrichment table 按钮。
按钮。 - 选择导出表格的文件位置和文件名。
默认文件扩展名为 .csv。
自动化¶
自动化用例也支持富集分析,基本命令语法为 enrichment analysis。你可以使用 organism 参数选择与查询基因关联的生物体,使用 geneID 参数选择带有基因符号的表格列。所有参数请参见这里。